نوترکیبی: نرعقیمی سیتوپلاسمی و برگرداندن باروری در گیاهان برتر
نرعقیمی سیتوپلاسمی(CMS) یک صفت توارث پذیر مادری در گیاهان برتر است که از بیان ژنهای جدید و اغلب شیمری واقع در ژنوم میتوکندری حاصل می شود. در بسیاری موارد، ژنهای غالب اختصاصی هسته، که تحت عنوان بازگرداننده باروری (Rf) نامیده می شوند، از بروز فنوتیپ نرعقیم ممانعت کرده و باروری را به گیاهان حامل ژنومهای CMS میتوکندریایی برمی گردانند. اگرچه ژنهای میتوکندریایی مرتبط با نرعقیمی، برای بسیاری از سیستمهای CMS شناسایی شده اند، یادگیری جنبه های مولکولی ژنهای برگرداننده و پروتئینهای کد شده بوسیله آنها، به تازگی شروع شده است.
برای سیستمهای برگرداننده باروری/CMS، دو سیستم پایه شناسایی شده اند: ساپروفیت و گامتوفیت. درسیستم ساپروفیت، ژنهای برگرداننده باروری، اثراتشان را در بافتهای ساپروفیت، مانند سلولهای tapetal یا سلولهای مادر گرده، بروز می دهند. بازگشت باروری می تواند منجر به تولید دانه گرده هاپلویید زایا شود اگرچه گامتهای فرد ممکن است حامل ژن بازگرداننده باروری نباشند. در مقابل، عقیمی گامتوفیتی در مرحله هاپلوئید پس میوزی بیان می شود و قابلیت حیات گامت، به سبب حضورآلل برگرداننده در آن، بوسیله ژنوتیپ گامت تعیین می شود.
نرعقیمی سیتوپلاسمی و بازگشت باروری، صفات مهم زراعی در گیاهانی چون ذرت، آفتابگردان، برنج و کلزا هستند که برای تولید بذور هیبرید در مقیاس تجاری لازم هستند. هیبریداسیون اجازه بهره بردن از هتروزیس را می دهد و بنابراین منجر به عملکرد بیشتر و پایداری بیشتر عملکرد می شود. علاوه بر تسهیل بهره برداری اقتصادی از سیستمهای CMS-Rf، مطالعه دقیق ژنهای Rf و CMS اطلاعات لازم برای افزایش آگاهی در مورد برهمکنشهای هسته، سیتوپلاسم را فراهم می کند.
2- مکانیسمهای نرعقیمی سیتوپلاسمی
2-1 چارچوبهای خواندن باز(Open Reading Frames) شناسایی شده بعنوان عامل نرعقیمی سیتوپلاسمی:
-توضیح: اگر یك توالی نوكلئوتیدی داشته باشیم به 3 نوع می توانیم آنرا بخوانیم یعنی یكبار از اول توالی شروع كنیم، باردوم یك نوكلئوتید را نمی خوانیم و ادامه می دهیم ، بارسوم دو نوكلئوتید اول را نمی خوانیم و از نوكلئوتید سوم شروع می كنیم . اگر در نظر بگیریم 61 كدون مختلف داریم اگر كدون ها تصادفاً پشت سر هم قرار گرفته باشند به صورت تصادفی باید بعدازهر 20 كدون یك كدون پایان وجود داشته باشد(3 كدون پایان داریم) . اما اگر یك توالی نوكلئوتیدی 600 تایی داشته باشیم نباید دربین آنها رمز پایان وجود داشته باشد. حال با توجه به این نكته و آنچه درباره انواع روشهای خواندن توالی نوكلئوتیدی گفته شد می توان بیان كرد اگر یك توالی نوكلئوتیدی مربوط به یك ژن در اختیار ما قرار گیرد، روشی از خواندن صحیح است كه به كدون پایان نرسیم ولی اگر به كدون پایان رسیدیم آن Frame صحیح نیست. پس به Frame صحیح خواندن كه در آن كدون پایانی وجود ندارد و راه برای خواندن توالی نوكلئوتیدی باز است Open Reading Frames) ORF) گویند و به دوFrame دیگر blocked reading frame گویند اگر در سه ORF ،Frame نداشته باشیم از روی آن توالی، پروتئین سنتز نمی شود .
چارچوبهای خواندن باز مرتبط با نرعقیمی سیتوپلاسمی، اغلب بیانگر ژنهای شیمری هستند که بنظر می رسد از حوادث نوترکیبی چندگانه که عمدتا در ژنهای میتوکندریایی شناخته شده و همچنین ناحیه در برگیرنده ’3 و ’5 و توالیهای با منشا ناشناس رخ می دهند، سرچشمه گرفته اند.
در برنج، ژن شیمری T-urf13 سیتوپلاسم T از طریق حوادث نوترکیبی مختلف تکامل یافته و شامل ناحیه در برگیرنده ’3 ژن 26S rRNA و همچنین نه اسید آمینه با منشاء ناشناخته است. نوترکیبی ها، T-urf13را مجاور ناحیه در برگیرنده ’5 ژن atp6 قرار داده اند که رونویسی T-urf13را ممکن می سازد. ژن T-urf13در فرادست یک ژن میتوکندریایی حفاظت شده،orf 221، قرار گرفته است. در CMS-S، دو ناحیه مجاور میتوکندریایی orf355 و orf77 مرتبط با نرعقیمی سیتوپلاسمی هستند. نسخه 1.6Kbحاصل از رونویسی orf355 و orf77 در میکروسپور گیاهان CMS-S حاضر و در میکروسپور گیاهان نربارور بازآوری شده از گیاهان CMS-S بوسیله موتاسیون میتوکندریایی، غایب هستند. orf77 حاوی سه ناحیه مشتق شده از لوکوس زیرواحد نهم ATP سنتاز (atp9) میتوکندریایی است. ویرایش نسخه های رونویسی orf355 و orf 77 و ژن atp9، پیش از این مطالعه شده است.
در Sorghum bicolor، چارچوب خواندن باز اختصاصی CMSسیتوپلاسم 9E، نیز یک ژن شیمری را نشان داده است. حداقل دو حادثه نوترکیبی، منجر به طویل سازی ژن coxI در ناحیه ’3 و’5 این نوع CMS میشوند. درپیازچه، ساختار CMS1، دو بار در ژنوم میتوکندری حاضر است، اما با نواحی’5 مختلف. دو توالی CMS1-1 و CMS1-2 شناسایی شده اند. بخشی از ساختار CMS1، از توالیهای ژنهای حیاتی atp9 و atp6 حاصل شده است. سه چارچوب خواندن باز orf 780, orf 744 وorf501 با استفاده از سه کدون آغازین که پروتئین های پیش بینی شده 27.5kDa, 29 kDa و 19kDa را به ترتیب رمزگشایی می کنند، پیش بینی شده اند. محصول ژن orf501 وابسته به اندازه پروتئین 18kDa(براساس تحرک الکتروفورزی) مرتبط با فنوتیپ CMS1 است.
در گیاه اطلسی، ژن pcf شامل ناحیه در برگیرنده و رمز کننده ’5 ژنatp9، توالی های کد کننده ژن coxII وهمچنین توالیهای چارچوب خواندن باز urfS است.
در کلزا، برای چهار نوع سیتوپلاسم نرعقیم (Polima, Ogura, Kosena وTournefortii-Stiewe)، چارچوبهای خواندن باز مرتبط با نرعقیمی شناسایی شده اند. در سیتوپلاسم Polima، فنوتیپ CMS بوسیله یک چارچوب خواندن باز جدید،orf224، که حداقل سه حادثه نوترکیبی را نیاز دارد، ایجاد شده است. orf224 شامل ناحیه در برگیرنده’5 orf158، و یا بعبارت دیگرorfB که اخیرا ثابت شده زیرواحد 8 ATPسنتتازF1F0 است، و بخشی از اگزون 1 پروتئین ریبوزومی S3 و توالی های نامعلوم است. در سیتوپلاسم Ogura، فنوتیپ CMS با یک قطعه 5/2 کیلوبازی NcoIمرتبط است. در این ساختار، سه چارچوب خواندن باز orf158, trnfM و orf138، شناسایی شده اند. در نهایت orf138 مرتبط با نرعقیمی بود. orf125 مرتبط با فنوتیپ CMS سیتوپلاسم Kosena، بسیار مشابه orf138 است. این دو چارچوب خواندن باز تنها از نظر دو اسید آمینه جایگزین و یک حذف 39-bp متفاوت هستند. برای Tournefortii-Stiewe CMS، یک ژن شیمری،orf193، که یک پروتئین پیش بینی شده 22.7kDa را رمز می کند، بخشی از توالی ژن atp6 را نشان داده و بعنوان ژن کاندید برای نرعقیمی مورد نظر است. اما، orf193 همراه با یکی از ژنهای اخیرا شناسایی شده atp9 نسخه برداری می شود و ممکن است بطور مداوم بصورت یک پروتئین شیمری 30.2 کیلودالتونی ترجمه شود.
درPhaseolus vulgaris، ناحیه منحصر به فرد pvs سیتوپلاسم نرعقیم CMS Sprite حاوی سه فرم orf98, orf97 و orf239 است. ناحیه pvs، که بوسیله ژن atpA در برگرفته شده است و توالی های ژنcob، هیچ همولوژی با DNA هسته ای، بجز با اینترون آلانین tRNA پلاستیدی و یک ناحیه کوتاه حاوی تکرارهای 5Kb در ذرت، نشان نمی دهند. توسعه ناحیه pvs، احتمالا ناشی از حوادث نوترکیب متفاوت است.
در CMS PET1 آفتابگردان، چارچوب خواندن باز orfH522 که مرتبط به نرعقیمی است در ناحیه ’3 ژن atpA موقعیت یابی شده است و حاوی 57bp ابتدایorfB) atp8) و توالیهای ناشناخته برای بخش باقی مانده است. orfH522 همراه با ژن atpA بصورت یک نسخه بزرگتر اضافی نسخه برداری شده است. برای سیتوپلاسمPEF1، الحاق 0.5Kb از توالی های نامعلوم در ناحیه ’3 ژن atp9 بعنوان عامل نرعقیمی شناخته شده است. حوادث نوترکیبی چندگانه ژنهای میتوکندریایی شناخته شده وهمچنین توالی های ناحیه نامعلوم، بنظر میرسد که چارچوب خواندن باز مرتبط با القای نرعقیمی در گیاهان برتر را ایجاد می کنند.
2.2 پروتئین های اختصاصی CMS و اعمال احتمالی آنها
برای بسیاری از سیستمهایCMS، پروتئین های مرتبط با CMS برای چارچوبهای خواندن باز مرتبط با نرعقیمی شناسایی شده اند. در دیگر حالات، بدون آگاهی در مورد چارچوب خواندن باز مرتبط، تنها حضور یا فقدان یک پروتئین در محصولات ترجمه سیستمهایCMS، در مقایسه با سیتوپلاسم بارور، مشاهده شده است.
در ذرت، یک پروتئین اضافی 13kDa شناسایی شده که در سیتوپلاسم T بیان شده و بوسیله ناحیه رمز کننده منحصر به فرد T-urf13 رمز می شود. پروتئین 13kDa بعنوان یک پروتئین میتوکندریایی مهم، که سبب حساسیت به T-toxin می شود، شناسایی شده است. T-toxin تولید شده بوسیله نژاد T پاتوژن Cochliobolus heterostrophus که سبب بلایت برگی ذرت می شود، با پروتئینی که در دیواره درونی میتوکندری با آرایش الیگومری جای گرفته، برهمکنش دارد. این امر سبب تشکیل سوراخ و نفوذپذیری دیواره می شود. پیوند پاتوتوکسین، سبب عدم انجام فسفوریلاسیون اکسیداتیو میتوکندریایی می شود. برای توقف رشد دانه گرده، مشخص شده که پروتئین 13kDa ممکن است با یک ماده اختصاصی پرچم بروشی مشابه T-toxin، برهمکنش یابد. برای CMS-C ذرت، یک پروتئین اضافی 17.5kDa شناسایی شده است. برایCMS-S، تغییرات پیچیده با هشت پروتئین اضافی که همگی بزرگتر از 42kDa هستند، مشاهده شده است.
درSorghum bicolor، محصولات ترجمه ژنوم میتوکندری بسیاری از سیستمهای CMS بررسی شده است. برای CMS میلو، یک پروتئین اضافی 65kDa، برای CMS 9E یک پروتئین اضافی و فقدان یک پروتئین38kDa شناسایی شده است. برای CMS IS II2 و همچنین ( MS M35-1(B به ترتیب پروتئین های اضافی 12kDa و82kDa شناسایی شده اند.
در اطلسی، محصول ژن pcf که مرتبط با سیتوپلاسم نرعقیم است بوسیله کاربرد آنتی بادیهای تولید شده برعلیه یک الیگوپپتید سنتتیک بررسی شده است. پروتئین 43kDa رمز شده بوسیله pcf، متحمل پردازش پس ترجمه در پایانه های N برای تبدیل به یک پروتئین 25kDa می گردد.
در کلزا، یک پروتئین 19kDa بعنوان محصول ژن orf138 که مرتبط با سیتوپلاسم Ogura است شناسایی شده است، و برای CMS نوع Juncea، یک پروتئین 32kDa مرتبط با فنوتیپ نرعقیم است. orf125 سیتوپلاسم نرعقیم Kosena، یک پروتئین 17kDa را رمز می کند. یک آنتی بادی رمز شده برعلیه ORF125، برای اثبات اینکه تجمع آنتی بادی در هیبریدهای با بازگشت باروری کاهش می یابد، استفاده شده است.
همچنین در Phaseolus vulgaris، آنتی بادی امکان تشخیص محصول ژن pvs-orf239 که با CMS مرتبط است، را فراهم کرده است. علاوه بر پروتئین پیش بینی شده 27.5kDa، یک پروتئین دیگر 21kDa شناسایی شده است که ممکن است محصول تخریب باشد. اما، برخلاف دیگر سیستمهای CMS، که پروتئینهای اختصاصی CMS همچنین در بافت رویشی بیان می شوند، پروتئین 27.5kDa درPhaseolus vulgaris تنها در سلولهای مادرگرده و میکروسپور در حال رشد یافت می شود.
در آفتابگردان، یک چارچوب خواندن باز جدید orfH522 در ناحیه در برگیرنده ’3 ژن atpA می تواند مرتبط با فنوتیپ CMSPET1 باشد. با استفاده از آنتی بادیهای اختصاصی برعلیه محصول ژنorfH522، مشخص شده که orfH522 پروتئین 16kDaرا رمز می کند که تنها تفاوت میان محصولات ترجمه اندامکی لاینهای بارور و لاین های نرعقیم است. پروتئین 16kDaبا جداره سلولی پیوند یافته و بیان آن بطور اختصاصی در پرچمهای هیبریدهای با بازگشت باروری کاهش یافته است. پروتئین 16kDa بنظر می رسد که در القای مرگ سلولی برنامه ریزی شده (Programmed Cell Death) پیش از بلوغ در سلولهای tapetum از طریق آزادسازی سیتوکروم C بوسیله میتوکندری سهیم است. با مقایسه پروتئین های رمز شده میتوکندریایی 28منبع CMS در آفتابگردان، نه منبع اضافی CMS شناخته شده اند که مکانیسم CMS مشابه با PET1 دارند. این امر بواسطه اینکه منابع CMS مشابهPET1، منشا متفاوتی دارند، شگفت انگیز است. این ژرم پلاسمهای نرعقیم سیتوپلاسمی بوسیله تلاقیهای متفاوت بین گونهای میان ARG1) H. phyllusargo)و H. neglectus (NEG1), H. exilis (EXI2), H. anomalus(بANO1) و دو زیرگونهH. praecoxبه نامهای (PRR1, PRH1) ویا بوسیله موتاژنزیز دو لاین نگهدارنده سیتوپلاسم PET1 تولید شده اند (MUT1 and MUT2). علاوه بر این، یکی از انواع CMS که بطور خود بخود ظهور می یابد (ANN10)، پروتئین 16kD را بیان می کند. تمامی این سیتوپلاسمهای مشابه PET1، سازمان مشابهی در لوکوس atpA نشان می دهند.
صرف نظر از این منابع CMS مشابهPET1، دیگر گروه های سیتوپلاسم CMSکه پروتئین های جدیدی را بیان می کنند شناسایی شده اند. ARG3 وRIG1 یک پروتئین اضافی 16.9kDرا نشان داده اند اما فاقد یک پروتئین17.5kDمشترک با دیگر سیتوپلاسمها بوده اند. ANN1 و ANN3 سه پروتئین اختصاصی 34.0, 16.9 و 16.3 را بطور مشترک بیان کرده اند. یک پروتئین 12.4kDمنحصر به PET2 و GIG1 بود. اگرچه هیچ چارچوب خواندن باز اختصاصی برای این پروتئینها هنوز شناسایی نشده است، اما ارزیابی های سطح DNA میتوکندریایی و همچنین الگوی متفاوت بازگشت باروری، گروه بندی منابع CMS را تایید می کند.
پروتئینهای مرتبط با CMS همچنین درگیاهان Allium schoenoprasum،Beta vulgaris Nicotiana،Triticum aestivum،Daucus carota و Vicia faba شناسایی شده اند.
گزارش شده که وزن مولکولی پروتئین های مرتبط با نرعقیمی سیتوپلاسمی از 12 تا 82 کیلودالتون می باشد. برای برخی از پروتئین های شناخته شده، حضور یا عدم حضور آنها تنها بواسطه مقایسه با سیتوپلاسم بارورمشخص شده است اگرچه همبستگی یا تایید مستقیم مشارکت آنها از طریق بررسی اثرات ژنهای برگرداننده بر بیان آنها تاکنون میسر نشده است.
3.2 عملکرد ژنهای PPR بعنوان ژنهای برگرداننده
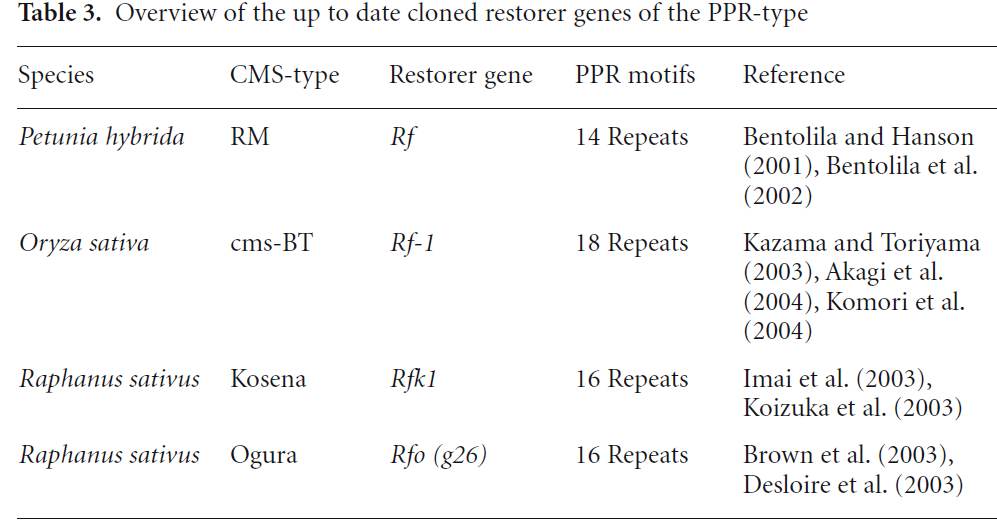
همه ژنهای ایزوله شده برگرداننده باروری، به استثنای ژن Rf2ذرت، متعلق به خانوادهای از ژنها هستند که حاوی یک موتیف تکراری pentatricopeptide) PPR) هستند(جدول3). پروتئینهای حامل موتیفPPR، آرایشی متوالی از تکرارهای یک موتیف نزول یافته 35 آمینواسیدی را نشان میدهند. درArabidopsis.thaliana، بوسیله یک خانواده ژنی 450 عضوی، پروتئینهای حاوی PPRبا تعداد تکرار 2 تا 26، رمز شدهاند. عمده این پروتئینها حدس زده میشود که مورد هدف میتوکندری یا کلروپلاست باشند. گمان میرود که پروتئینهای PPR بطور اختصاصی با RNA در اندامکها برهمکنش دارند و در پردازش یا ترجمه RNA نقش بازی میکنند.
نخستین ژن برگرداننده باروری از نوع PPR، از گیاه اطلسی جداسازی و کلون شد. براساس تکنیکهای مارکر مولکولی، ناحیه ژنومی حاوی ژن برگرداننده، به یک BIBAC با اندازه 37.5 kbمحدود شده بود. سپس روشهای ترنس ژنیک برای شناسایی ژن برگرداننده از میان ژنهای کاندید مورد استفاده قرار گرفتند. ژن برگرداننده 592 اسید آمینه را کد میکند و بواسطه یک سیگنال متوالی از میتوکندری کنترل میشود. Rf-PPR592 باروری را به گیاهان ترنس ژنیک حامل سیتوپلاسم اطلسی برگردانده است. فراوانی پروتئین PCFمرتبط با CMS، بطور قابل ملاحظهای در این گیاهان کاهش مییابد. Rf-PPR592حاوی 14 کپی از موتیف pentatricopeptideاست و شامل 87 درصد از ناحیه رمز گذار میشود. لوکوس Rf در اطلسی ساختار ژنومی پیچیدهای را نشان میدهد و شامل یک ژن ثانویه (Rf-PPR591).PPR رمز گذار یک پروتئین 591 اسید آمینهای با عملکرد نامعلوم است. در مقایسه با Rf-PPR592، ژن همولوگ در لاینهای rf/rfدارای یک حذف در ناحیه پروموتر بوده و متفاوت از نظر توالی پیش بینی شده اسید آمینه است.
در برنج، نرعقیمی cms-BT (cms-bo)بواسطه حضور ژن غیرنرمال (B-atp6) atp6در ژنوم میتوکندری است. B-atp6بصورت یک نسخه 2 کیلوبازی رونویسی میشود که علاوه بر atp6، حاوی یک توالی منحصر به فرد از orf79 در ناحیه '3 ژن atp6است. در حضور ژن برگرداننده Rf-1، دو نسخه 1.5 و 0.45 kbبوسیله پردازش نسخه 2.0 kbبوجود میآیند. آنالیز توالی cDNAثابت میکند که B-atp6-RNA پردازش شده بطور موثری بصورت N-atp6-RNAویرایش شده است. ژن Rf-1بنظر میرسد که در پردازش سهیم است وهمچنین بر فرایند ویرایش پس از ترجمه atp6تأثیر دارد. برخی گروههای تحقیقاتی اقدام به کلون کردن ژن Rf-1بوسیله ترکیب یک استراتژی کلونینگ براساس نقشه یابی و یک رویکرد مبتنی بر ژن کاندید کردهاند. نخستین بار در سال2003 ژن PPR8-1، رمزگذار یک پروتئینPPR، بعنوان ژن کاندید برای Rf-1، شناسایی شد. با استفاده از یک رویکرد ترنس ژنیک، ثابت شد که این نسخههای ژنی در پردازش نسخههای atp6نقش دارند. پروتئین کد شده بوسیله PPR8-1، به تشکیل 0.45 kb RNAاز رونوشت ژن B-atp6، به همان نحو که در موردRf-1 حدس زده میشود، کمک میکند. اما، این مسئله که آیا معرفی (واردسازی) PPR8-1سبب باروری گیاهان باززایی شده میشود یا خیر، بررسی نشده است. PPR791 (Rf-1)که متعاقب یک استراتژی نقشه یابی دقیق کلون شده است همانند PPR8است. ژن Rf-1حدس زده میشود که یک پروتئین 791 اسید آمینهای، حاوی 16 موتیفPPR، را رمز میکند که از این بین، 14 موتیف پشت سرهم قرار دارند. به سبب حذف 1-bpدر ناحیه رمز گذار توافقی آلل مغلوب (rf-1)که منجر به frame shiftو تولید زودهنگام کدون توقف میشود، این ژن یک پروتئین کوتاه شده 266 آمینو اسیدی، را رمز میکند. حذف 574 bpحاضر در ناحیه'3توالی رمز کننده، احتمالاً بر عملکرد ژن برگرداننده تأثیری ندارد.
در کلزا یک ژن برگرداننده از نوع PPRبرای Ogura-CMSو Kosena-CMSایزوله شده است. ژن Rfoحاصل ازRaphanus میتواند نرباروری را به کلزای حاوی سیتوپلاسم Ogura برگرداند. برخلاف Rfاطلسی، Rfoبر نسخههای متناظر با ژن میتوکندریایی مرتبط با نرعقیمی سیتوپلاسمی،orf138 ، تأثیری ندارد. Rfoدر میزان ترجمه یا پس ترجمه تأثیر داشته و منجر به کاهش محصول ژن orf138در برگها و گلها میشود. کلونینگ ژن Rfoبواسطه کاربرد synteny (روابط ژنومی) میان Raphanusو Arabidopsisتسهیل میشود، اگرچه Arabidopsis یک ژن PPRمتناظر با ژن برگرداننده Rfoندارد. ژنg26، رمز کننده یک پروتئین 687 اسید آمینهای با یک توالی پیش بینی شده هدف گذار میتوکندریایی، بعنوان ژن کاندید برای Rfoشناسایی شده است.
ژنهای در برگیرنده، g24 و g27، حاوی موتیفهای چندگانه PPR هستند، اما هر دو فاقد سومین تکرار g26 هستند. پروتئینهای پیش بینی شده رمز شده بوسیله این سه ژن از نظر طول مشابه هستند، g24p و g27p به ترتیب687 و654 اسید آمینه دارند در حالیکه محصول ژن Rfo(g26p) دارای687 اسید آمینه است. ترنسفورماسیون با استفاده از یک کلون حاوی g26، سبب تولید گیاهان باززایی شده بارور گردید. همچنین سه ژنPprA،PprB :PPR و PprC بر تربچهBAC-clone 64 یافت شدهاند. آنالیز توالی PprC پیشنهاد میکند که PprC یک ژن کاذب است. بطور خلاصه، Rfo احتمالاً متناظر با PprA یا PprB و یا هردو است.
آنالیز CMS میتوکندریایی تربچهKosena نشان میدهد که Kosena حامل orf125 است که یک پروتئین 17-kDa را رمز میکند و حاوی توالی همولوگ با orf138، بجز دو آمینو اسید جایگزین و یک حذف 39-bp در ناحیه رمز کنندهorf138، است. تجمع ORF125 و ORF138 مرتبط با فنوتیپ CMS درBrassicaاست. ژن Rf بیان پروتئین در سطح ترجمه را تنظیم میکند. دو لوکوس هستهای Rfk1 و Rfk2، با آللهای غالب قادر به برگرداندن باروری به تربچه Kosena دارای سیتوپلاسم نرعقیم هستند. اگرچه میزان رونویسی orf125 در حضور آلل غالب Rfk1 تغییر نمیکند، تجمع پروتئین ORF125 بطور قابل توجهی کاهش مییابد. براساس استراتژی کلونینگ موضعی، ناحیه لوکوس برگرداننده باروری Rfk1 محدود به 43-kb در تربچه Kosena است که بوسیله چهار کلون lambda و یک کلون کاسمید پوشانده شده است. برای شناسایی Rfk1، زیر کلونهای حاوی ناحیه 43-kb از طریق ترنسفورماسیون با Agrobacterium بدرون لاین سیتوپلاسم نرعقیم B. napus وارد شدند. orf687 که 687 اسید آمینه با وزن مولکولی 76.5 kDa را رمز میکند بعنوان Rfk1 شناخته شده بود. آلل مغلوب حاوی 11 باز جایگزین است. پنج باز جایگزین، که سبب تولید چهار اسید آمینه جایگزین میشوند، همگی در ناحیه تکرارهای PPR واقع شدهاند. توالی پروتئین ORF687 رمز شده بوسیله Rfk1 مشابه پروتئین g26p است. بنابراین، اگرچه تفاوتهایی از نظر شاخصههای سیتوپلاسم نرعقیم میتوکندریایی میان سیتوپلاسم Kosena و Ogura تربچه وجود دارد، بازگشت باروری به هر دو سیستم میتواند بوسیله ژنهای رمز کننده پلی پپتید یکسان انجام شود.
این امر که بسیاری از ژنهای برگرداننده، موتیفهای PPR را نشان میدهند بطور قابل توجهی ایزوله کردن ژنهای کاندید برای ژنهای برگرداننده را در دیگر گونهها تسهیل میکند.
4. نتیجه گیری
عملکرد میتوکندری مشخصاً برای رشد دانه گرده حیاتی است. سیستمهای CMS و ژنهای متناظر برگرداننده باروری، امکانات مناسبی برای مطالعه نقش میتوکندری در رشد گرده و برهمکنش هستهها و میتوکندری در این رابطه را ارائه میکنند. اگرچه عوامل نرعقیمی سیتوپلاسمی با جزئیات در سطح مولکولی، مطالعه شدهاند ایزولاسیون ژنهای برگرداننده تنها شروع شده است. شناسایی ژنهای برگرداننده متعلق به خانواده ژنی PPR ممکن است احتمال پیشرفت بهتر در کلونینگ آتی ژنهای برگرداننده را بوسیله ترکیب کلونینگ برپایه نقشه یابی با رویکردهای ژن کاندید را میسر سازد. با این همه، تنها آینده نشان خواهد داد که آیا تمامی ژنهای برگرداننده متعلق به یک نوع مشابه هستند و یا اینکه دیگر انواع ژنهای برگرداننده مشخص خواهند شد. برای این ژنها، فرآیند کلونینگ طولانیتر خواهد بود و سیستمهای ترنس پوزون- تگ گذاری، بعنوان جایگزینی برای استراتژی کاربرد کلونینگ برپایه نقشه یابی، برای تمامی گیاهان میسر نیستند. اگرچه چارچوبهای خواندن باز مرتبط با CMS و پروتئینهای رمز شده شناسایی شدهاند عملکرد و نقش این پروتئینها، صرف نظر از پروتئین T-URF13 ذرت، هنوز معلوم نشده است. ایزولاسیون و مطالعه عملکرد ژن برگرداننده ممکن است فهم بهتر فرآیندهای دخیل را میسر سازد. اما، بطور کلی بنظر میرسد که هر گونه اختلال در عملکرد میتوکندری، برای فرآیند به شدت انرژی خواه رشد دانه گرده در گیاهان برتر، مرگبار خواهد بود.
برای آنالیز مولکولی نرعقیمی سیتوپلاسمی و بازگشت باروری، مارکرهای مولکولی مناسبی ارائه شده است که انگشت نگاری هیبریدها و ارزیابی خلوص بذور هیبرید بمنظور کاربردهای تجاری، را برپایه استراتژیهای مبتنی بر PCR میسر میسازد.
این مطلب، ترجمهای از مقاله:
"Recombination:Cytoplasmic male sterility and fertility restoration in higher plants"
نوشته Renate Horn(2006)بود، جهت دانلود متن اصلی مقاله، به همراه منابع آن، بر روی لینک زیر کلیک فرمایید:
http://s1.picofile.com/file/7545474408/CMS_Rf.pdf.html


 مقاله نقشه یابی مولکولی صفات گیاهان زراعی
مقاله نقشه یابی مولکولی صفات گیاهان زراعی توی پنجره دوجداره
توی پنجره دوجداره باردهی درختان ميوه به چه عواملی بستگی دارد
باردهی درختان ميوه به چه عواملی بستگی دارد آموزش جدا کردن پاجوش های آلوئه ورا
آموزش جدا کردن پاجوش های آلوئه ورا اطلاعات کامل درخت گلابی در ایران
اطلاعات کامل درخت گلابی در ایران معرفی گل درختچه ها آپارتمانی زیبا سازی باغچه
معرفی گل درختچه ها آپارتمانی زیبا سازی باغچه روش کاشت پیاز سنبل
روش کاشت پیاز سنبل شيره خام شيره پرورده درخت چیست
شيره خام شيره پرورده درخت چیست گیاه پاندانوس
گیاه پاندانوس  راهنما استاندارد های کشاورزی کاردانش
راهنما استاندارد های کشاورزی کاردانش کاکتوس کلاه اسقف
کاکتوس کلاه اسقف درخت آقطی سیاه
درخت آقطی سیاه